
中大新聞網訊(通訊員駱觀正)自從在真核生物DNA上重新發現N6-deoxyadenosine(6mA)修飾以來,研究人員對其能否成為新的表觀遺傳標記充滿期待。隨著研究的進展和新方法的開發,研究者們聲稱6mA存在于各種真核生物中,包括植物、脊椎動物,甚至哺乳動物。然而,現有方法的不足限制了6mA的精確定位和功能解釋。近年來關于6mA是否存在于哺乳動物中以及其能否成為新的遺傳學標記的重要性問題引起了廣泛的爭論。因此,開發一種敏感和可靠的檢測方法來確定6mA的存在以及基因組中真正的6mA位點的精確定位至關重要,特別是對于6mA水平非常低的生物。
中山大學生命科學學院駱觀正教授長期專注于表觀遺傳相關研究,前期工作發現了6mA在真核生物中是一種可能的新型表觀遺傳標記(Cell, 2015),并對其在高等生物中潛在的生物學功能進行了總結和展望(Nat Rev Mol Cell Bio, 2015; Nat Struc Mol Bio, 2017)。近日,駱觀正教授團隊報道了一種基于新的檢測原理的全基因組6mA檢測新方法MM-seq (Modification-induced Mismatch Sequencing),并利用該方法評估了6mA在細菌、綠藻和哺乳動物細胞中的基因組位置,揭示了人類不同細胞類型擁有非常有限的6mA位點,這些位點零星分布在不同的基因組定位上。敲除RNA m6A甲基轉移酶METTL3后,基因組DNA中可檢測到的6mA位點進一步減少。
該研究中發現在雙鏈DNA中,6mA特異性抗體可以識別嵌入在DNA雙鏈上的修飾腺嘌呤。在研究了6mA的化學性質后,研究者推測6mA的存在減弱了堿基的堆積,并使局部DNA雙鏈不穩定。研究者通過高錳酸鉀足跡法證實了雙鏈DNA上的N6 -脫氧腺苷促進了類似錯配結構的產生,其機制可能是通過DNA呼吸或堿基翻轉。這種獨特的類錯配結構允許6mA被6mA特異性抗體捕獲,隨后被特定的核酸酶或化學裂解反應識別。隨后,根據這一原理開發了一種全新的6mA定位方法MM-seq (modified -induced Mismatch Sequencing)(圖1)。
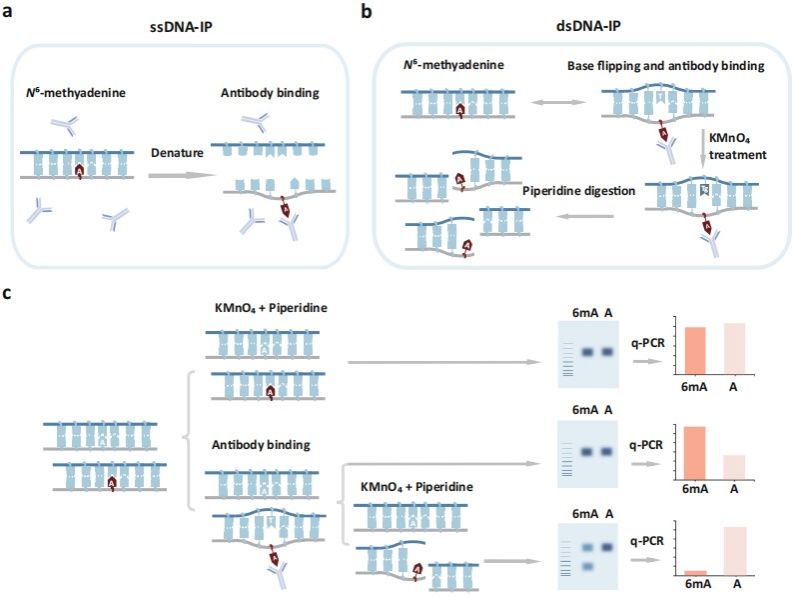
圖1. KMnO4足跡法檢測6mA誘導的錯配樣結構
鑒于MM-seq的高精度,研究人員隨后將該方法應用于6mA水平較低或常規測序方法無法檢測到的哺乳動物細胞當中。結果發現多種人類細胞類型其基因組DNA上含有非常有限的6mA位點,這些位點隨機分布在不同的基因組位置上,不同細胞中的位點重復性極低。考慮到雖然這些單堿基6mA位點非常罕見,但這些有限的位點可能富集在特定的基因組區域,并發揮重要的調控作用。因此,進一步研究了6mA位點是否有聚集在特定區域的趨勢,特別是在基因組或線粒體DNA的重復元件(REs)上。結果發現傳統IP鑒定的6mA峰在REs處富集,然而,MM-seq鑒定的位點在此并無富集,提示之前研究中發現的6mA富集在重復元件的現象很可能是因為方法的假陽性造成的。
針對哺乳動物中6mA含量極低的特征,最近有研究提出6mA是由于RNA上修飾堿基經過消化代謝,被DNA聚合酶隨機整合進DNA的觀點。為了驗證這一觀點,研究人員進一步敲除RNA m6A甲基轉移酶METTL3后再進行MM-seq檢測。結果表明,基因組DNA中可檢測到的6mA位點進一步減少,該研究結果支持了哺乳動物基因組中有限的6mA可能源于RNA m6A的代謝副產物。
綜上,該研究不僅提供了一種全新的方法來全面和準確地繪制多個物種中6mA的分布,而且還將加深對罕見DNA修飾的理解。近日,該研究成果“High-precision mapping reveals rare N6-deoxyadenosine methylation in the mammalian genome”在Cell Discovery雜志上發表,陳麗倩博士(現為廣東省人民醫院博士后)和張璋副教授為共同第一作者,駱觀正教授為通訊作者,課題組成員陳鴻萱、奚劍飛、劉學弘、馬冬曌、鐘宇浩、黃文慧、陳濤參與了該工作。香港大學Daniel W. Mak、中山大學公共衛生學院(深圳)的陳琦和陳耀慶對該論文提供了幫助。文中所涉及到的方法已申請發明專利。