
中大新聞網訊(通訊員李佳璇)擴增子捕獲 (Amplicon capture) 是一種經濟高效的分子系統學數據收集方法。該方法首先利用通用擴增引物集從目標生物類群的代表物種中擴增大量的目標PCR片段 (即擴增子);然后在這些片段的兩端添加生物素化的接頭,并用生物素標記的接頭引物對片段進行擴增,得到大量含有生物素標記的擴增子探針;接著就可以利用這些探針從DNA文庫中“釣”取特定區域并進行高通量測序 (圖1)。在擴增子捕獲實驗中,擴增子探針的數量和組成可以根據項目需求靈活調整,制備周期短,實驗成本低,捕獲得到的數據完整,因此該方法廣泛適用于從種級至綱級的系統發育基因組學研究。
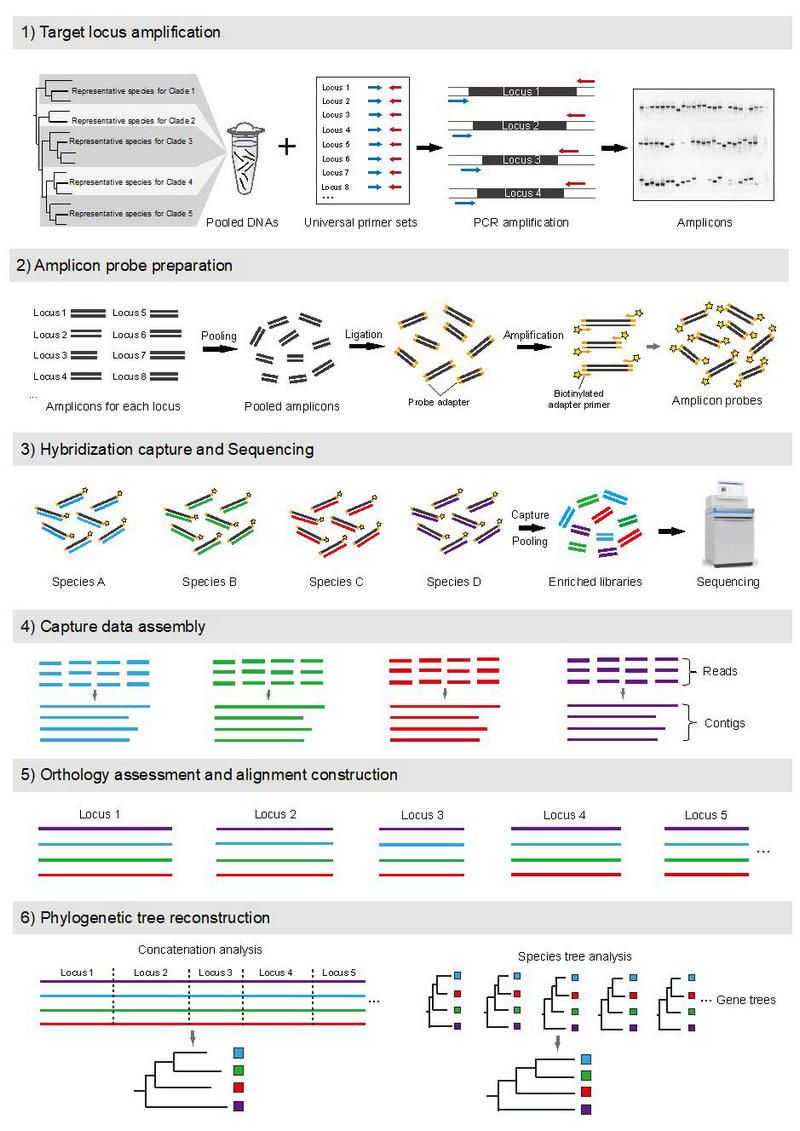
圖1. 擴增子捕獲用于系統發育基因組學研究的一般工作流程
開展擴增子捕獲實驗的關鍵在于擁用大量類群通用的擴增引物集,但目前這些通用的擴增引物資源卻相當有限,僅在脊椎動物、甲蟲和鱗翅目昆蟲中有少量的積累,這極大限制了擴增子捕獲的廣泛應用。目前國際數據庫中動物的基因組數據正在快速積累,覆蓋了大多數后生動物類群,這為通用擴增引物的開發提供了重要的數據來源。然而,大規模分析大量物種的基因組數據并進行引物開發需要豐富的基因組數據分析經驗和引物設計經驗,對絕大多數研究者而言是一項巨大的挑戰。針對這一問題,中山大學生命科學學院張鵬團隊開發了一款名為UPrimer的通用擴增引物設計程序。它能夠以基因組數據為輸入,全自動化的為給定生物類群大規模開發通用擴增引物集,助力擴增子捕獲實驗在相關生物類群中開展。
UPrimer采用Python語言編寫,包含兩大模塊,涵蓋了通用擴增引物開發的全部過程。與其他的通用引物設計程序相比,UPrimer有兩個獨創之處:一是具備全自動化的引物開發模式。研究者僅需要輸入一行簡單的命令,UPrimer就能自行分析基因組數據并輸出目標生物類群的通用擴增引物表。該表可直接用于引物合成以及后續的擴增子捕獲實驗。二是引物設計采用巢式PCR引物設計策略,即設計兩對引物擴增目標區域。該策略能夠大大增加基因片段的擴增成功率 (>90%),顯著提升擴增子探針制備的效率。
借助UPrimer,研究人員為21個后生動物類群開發了擴增通用引物集,覆蓋了節肢動物、軟體動物、刺胞動物、扁形動物、棘皮動物以及脊椎動物等主要多細胞動物分類單元,每套引物集包含約1,000個基因片段。為了驗證UPrimer開發引物的擴增性能,研究團隊選取了六個后生動物代表類群進行PCR擴增測試。結果表明,無論在亞目、目、綱還是亞門級的分類水平,UPrimer設計的通用引物集均能高特異性的擴增出目標基因片段,擴增成功率均超過90% (圖2),這表明UPrimer開發的引物集非常易于使用,完全能滿足擴增子捕獲探針制備的要求。
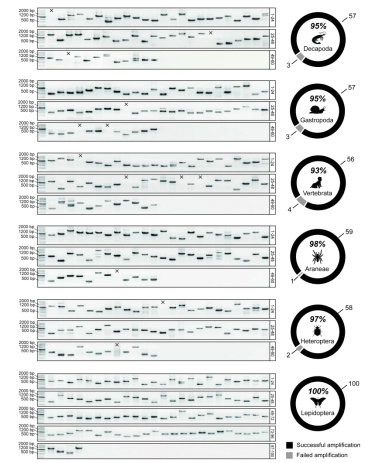
圖2. 六個后生動物類群(十足目、腹足綱、脊椎動物亞門、蜘蛛目、異翅亞目、鱗翅目)引物集的PCR擴增試驗結果
擴增子捕獲與近年來廣泛采用的基于商業合成探針的AHE、UCE捕獲測序相比,在探針制備成本、探針制備周期、捕獲片段的平均長度上均有明顯優勢(表1)。Uprimer引物自動化開發功能的實現,讓非生物信息學專家也能輕松地通過基因組分析獲取所需的通用引物集,進而借助擴增子捕獲以低成本、快速、便捷的方式獲取基因組級別的分子數據。隨著更多類群通用性引物集被開發出來,擴增子捕獲在未來的系統發育基因組學研究中將具有廣泛的應用前景。
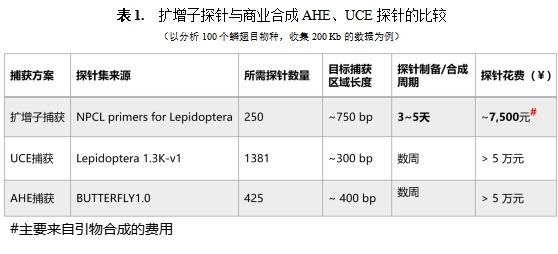
表1. 擴增子探針與商業合成AHE、UCE探針的比較
(以分析100個鱗翅目物種,收集200 Kb的數據為例)
近日,該成果以“UPrimer: A Clade-Specific Primer Design Program Based on Nested PCR Strategy and Its Applications in Amplicon Capture Phylogenomics”為題發表在國際進化生物學的權威期刊Molecular Biology and Evolution上。中山大學生命科學學院張鵬教授和梁丹副教授為該項工作的共同通訊作者,原博士后李佳璇 (現于暨南大學生命科學技術學院工作) 為該文第一作者。此項研究得到了國家自然科學基金等項目資助。
論文鏈接:https://academic.oup.com/mbe/article/40/11/msad230/7313555