
中大新聞網訊(通訊員楊建華)逆轉錄轉座子(Retrotransposons)在大多數物種中都普遍存在,能夠通過“復制-粘貼”機制進行大量擴增,在人類基因組中占比超過35%。逆轉錄轉座子可以為創造新的功能基因提供原料,是進化多樣性的最主要來源之一。它們能引發現有基因序列的改變,或充當新基因的啟動子,影響基因選擇性剪接,甚至促使新基因形成。具有表達潛能的逆轉錄轉座子對人體生理、組織分化、癌癥的發生發展具有深遠的影響,且被認為在哺乳動物大腦進化中扮演重要角色。然而,現有的逆轉錄轉座子檢測方法存在局限,需要已知確切序列,然后進行有監督的同源搜索。研究通常僅限于幾種經典的LINE/SINE元件,缺乏全面系統的基因組尺度探索。因此,各種類型的逆轉錄轉座子在全基因組的精確分布圖譜、生物生成、動態變化、潛在功能和作用機制仍有待闡明。
近日,中山大學生命科學學院楊建華/屈良鵠教授團隊開發了在全基因組檢測逆轉錄轉座子的新計算方法retroSeeker。通過將逆轉錄轉座子事件的生物學模型(復制-粘貼)映射到比較基因組學數據(gap和fill)中,retroSeeker算法能夠準確地識別任何物種基因組中的新逆轉錄轉座子。具體而言,親本基因的存在產生了一個“填充”(fill)區域,而“復制-粘貼”過程則在新的基因座產生了一個“間隙”(gap)區域,通過將物種間的基因組進行兩兩比對,可以復現這些“填充”和“間隙”區域以獲得候選的轉座子區域 (圖1A)。此外,根據逆轉錄轉座的TPRT(target-site primed reverse transcription)機制,逆轉錄轉座子的復制往往還需要借助其poly(A) 尾巴,且在插入新基因座后,逆轉錄轉座子兩側還會形成標志性的TSD(target site duplications)序列。因此,一旦retroSeeker確定了潛在的逆轉錄區域,它會立即利用動態規劃算法對側翼的TSD和poly(A)序列進行搜索和評分,最終獲得高置信度的逆轉錄轉座子候選 (圖1A)。通過模擬數據和真實數據的測試,retroSeeker算法表現出高度特異性、高靈敏度和快速的運行速度。(圖1B-E)。
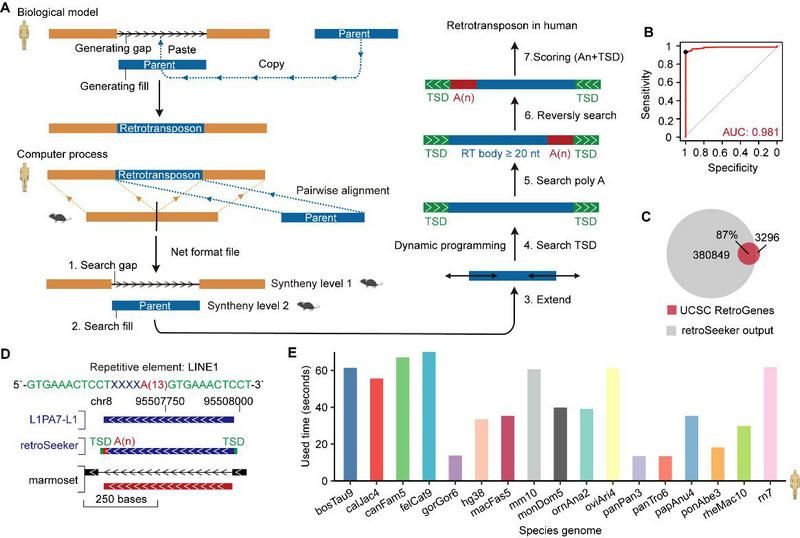
圖1 鑒定逆轉錄轉座子的新計算方法retroSeeker概覽
應用retroSeeker算法于人、小鼠和果蠅基因組,retroSeeker鑒定了大批新類型的逆轉錄轉座子,并解碼它們的生物發生、表達、進化和潛在功能。由于retroSeeker可對轉座子的插入位置進行單堿基精度的鑒定,研究團隊發現大多數新的逆轉錄轉座子表現出特定的L1內切酶切割基序,其中一些基序精確地位于插入位點上游的10個核苷酸(圖2A)。結合大規模的基因表達數據,發現大量的候選新功能基因可能通過逆轉錄轉位機制產生,例如,一些蛋白編碼基因本身不具備編碼miRNA的能力,但是通過逆轉錄轉座形成轉座子后,能夠進一步產生新的miRNA基因(圖2B)。有趣的是,研究團隊還發現了組蛋白基因、線粒體基因和vault RNA基因通過逆轉座子機制產生了新類型的逆轉錄轉座子(圖2C-E)。結合ENCODE/CCLE大規模的組織/癌癥表達數據,作者進一步闡明了逆轉錄轉座子的組織特異性表達,并證明了它們在各種癌癥類型中的普遍表達。最后,應用retroSeeker算法于更多物種的基因組,揭示了逆轉錄轉座子的復雜進化模式,并發現了許多物種特異性的逆轉錄轉座子事件(圖2F)。綜上,該研究開不僅鑒定了逆轉錄轉座子在各物種基因組的精確分布圖譜,也為進一步闡明逆轉錄轉座子的特性及其在生理和病理過程中的潛在作用提供了新算法。
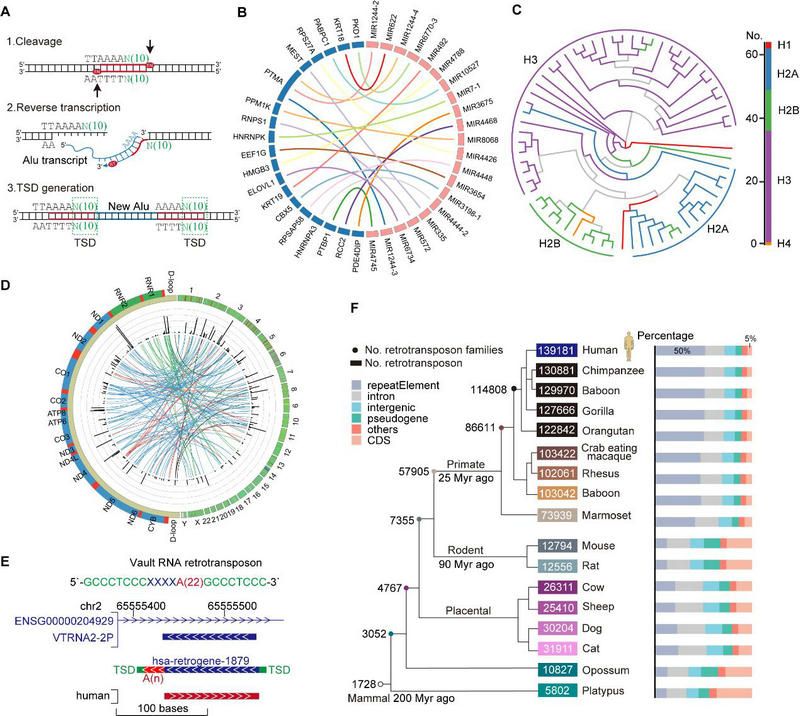
圖2 應用retroSeeker解碼逆轉錄轉座子的生物發生、表達、進化和潛在功能
該研究以“RetroSeeker Reveals the Characteristics, Expression, and Evolution of a Large Set of Novel Retrotransposons”為題發表在Advanced Biotechnology。中山大學生命科學學院楊建華教授、屈良鵠教授為該項工作的共同通訊作者,博士研究生黃鈞鴻為本文的第一作者。該研究得到了國家自然科學基金、國家重點研發計劃等項目資助。
論文鏈接:https://link.springer.com/article/10.1007/s44307-023-00005-5