
中大新聞網訊(通訊員唐振超)一種藥物不能滿足多種復雜疾病的治療,很多時候需要兩種或多種藥物共同治療多種疾病。但是當人們服用一種以上的藥物時,藥物之間的相互作用可能會對人體造成不良影響,這種現象稱為藥物相互作用(DDI)現象,因此在服用多種藥物之前確認DDI至關重要。傳統上,DDI的檢測是通過廣泛的生物學或藥理學分析進行的。然而,這個過程是費時費力的,況且藥廠也沒有這么多人力物力來驗證所有上萬種藥物之間的交互作用,因此深度學習方法可以作為一種低成本但有效的替代方法,通過識別已知DDI的模式來預測潛在的DDI。藥物相互作用在體內會引發意想不到的藥理作用,并且通常具有未知的因果機制。雖然現在已經開發了深度學習方法以更好地理解DDI,然而精確的預測DDI仍然是一個挑戰。具備可泛化的DDI預測比源域預測更接近現實場景。
中山大學智能工程學院智能醫療研究中心主任陳語謙教授團隊根據“藥物是一個由不同官能團/化學子結構組成的實體,子結構決定了藥代動力學和藥效學特性,并影響著相互作用”提出了DSIL-DDI方法,學習領域不變的子結構相互作用,以提高模型的泛化能力。DSIL-DDI將子結構相互作用視為DDI的域不變。在使用圖神經網絡(GNN)提取子結構后,子結構交互模塊用于學習領域不變子結構交互模式。對于提出的子結構交互模塊,其模擬子結構中屬性的交互。為了學習領域不變表示,DSIL-DDI中設計了一個額外的損失函數,可以從不相關的子結構相互作用中去除噪聲。對于一對藥物,模塊會輸出DDI表示。該表示包含與此DDI事件對應的最重要的子結構交互模式。將表示提供給分類器以獲得此DDI的類別。對于分布外(OOD)DDI預測,計算陌生域上的DDI表示(無需重新訓練),然后按指定數量的類別對這些表示進行聚類。該工作可以總結在圖1中。
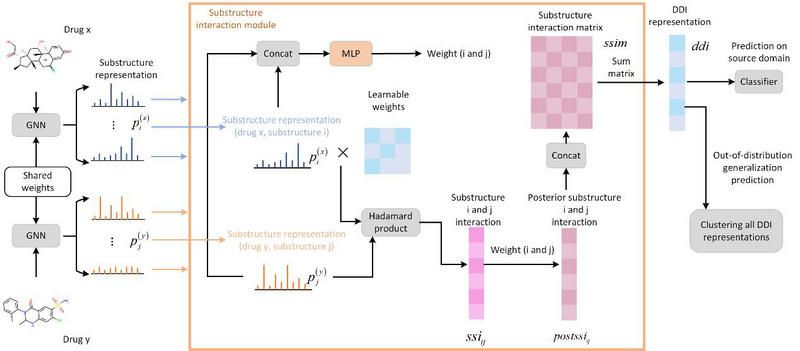
圖1 DSIL-DDI的網絡架構。給定輸入藥物對,GNN 提取其子結構表示;子結構相互作用模塊計算所有子結構的相互作用,聚合子結構相互作用矩陣得到DDI event的領域不變表示。
根據兩個基準評估DSIL-DDI:Drugbank和Twosides。實驗分為三種情況:transductive設置(熱啟動,測試集中的所有藥物都出現在訓練集中)、inductive設置(冷啟動,測試集中包含訓練集中不存在的新藥物)和OOD泛化。但是,前兩個設置共享相同的數據集/域。對于OOD泛化,需要對不熟悉的域進行預測。實驗結果表明:(1) DSIL-DDI 在兩種設置上都達到了良好的性能;(2)對于子結構相互作用,DSIL-DDI可以呈現出與藥物化學一致的分析,證明其可解釋性;(3)DSIL-DDI可以學習域不變表示,這證明它對OOD預測具有泛化性。
為了驗證模型在判別式預測中的可解釋性,該工作通過從藥物化學的角度研究這些藥物子結構的組合如何導致潛在的DDI,來證明DSIL-DDI的可解釋性。根據注意力權重索引藥物對中貢獻最大的子結構對(子結構相互作用)。然后將這些子結構相互作用與現有文獻進行比較。如圖2所示,展示了3個DDI事件并可視化了它們的子結構對。
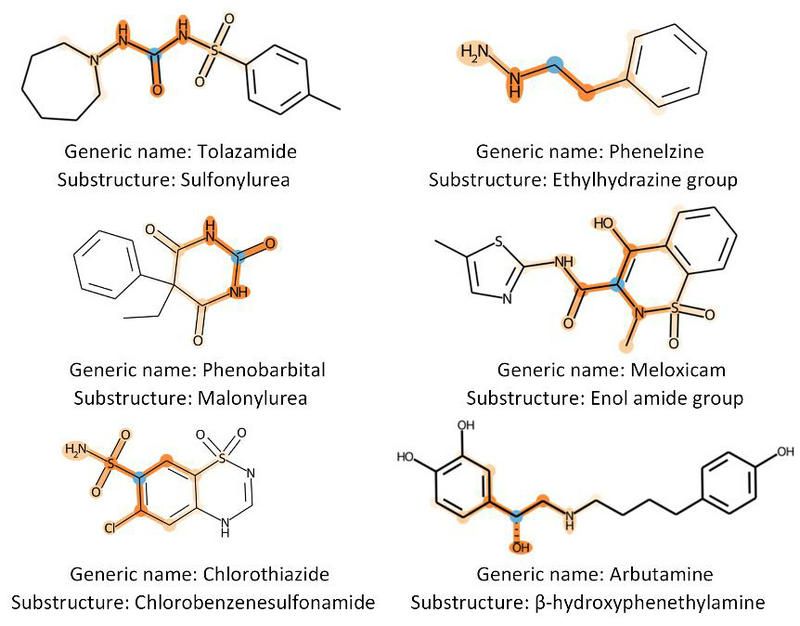
圖2 子結構相互作用的可解釋可視化。
研究成果于2023年2月在國際著名期刊IEEE Transactions on Neural Networks and Learning Systems上發表,題目為“DSIL-DDI: A Domain-Invariant Substructure Interaction Learning for Generalizable Drug–Drug Interaction Prediction”。中山大學智能工程學院陳語謙教授為該文通訊作者,智能工程學院博士生唐振超為第一作者。該項研究受到國家自然科學基金項目的支持。