
中大新聞網訊(通訊員張鵬)重建快速分化類群的演化歷史一直都是系統發育基因組學研究所面臨的挑戰之一。近年來的研究發現,解決這類挑戰不僅需要分析基因組級別的數據,還需要同時分析不同類型的數據,如編碼數據和非編碼數據。目標序列捕獲技術是收集上述數據的有效策略。目前大多數的序列捕獲方法均是使用定制的探針集,這需要研究人員根據目標類群的基因組序列設計探針,隨后由公司合成這些探針。然而,定制合成一套探針集的整個過程既耗時(至少6周)又昂貴(每個捕獲反應花費約1300元人民幣),更重要的是,許多非模式生物類群缺少可用的基因組資源來設計探針。
對此,中山大學生命科學學院張鵬教授團隊開發了一種基于自制全長cDNA探針的序列捕獲方法 (Full length cDNA capture; FLc-Capture) 來同時收集編碼和非編碼的系統發育基因組學數據(實驗流程見圖1,數據分析流程見圖2)。FLc-Capture方法只需要研究者從目標類群中挑選一個常見物種作為探針制備物種,并從中提取高質量的mRNA,然后以此為模板合成生物素化的全長cDNA作為捕獲探針。這種探針制備策略直接跳過了探針設計與合成的步驟,使研究人員可以在不需要基因組數據和商業探針合成的情況下,為任意一類生物制備一套捕獲探針集,具有高度的靈活性,且特別適合非模式生物的研究。利用該方法制備一套全長cDNA探針的總時長不超過3天,每個捕獲反應的花費不足10元,這比定制合成一套探針集更經濟、省時。為了證明FLc-Capture方法的實用性,研究團隊利用榕蛇的mRNA制備了全長cDNA探針,然后利用這些探針成功地從24個游蛇物種和12個遠緣蛇類樣本中捕獲到數千條編碼和非編碼序列。所有測試樣本的平均捕獲效率可達到35%,不亞于商業合成探針的捕獲效果。基于這些捕獲數據,研究者構建了兩個系統發育基因組學數據集來研究游蛇科的系統發育關系,其中數據集1包含了1,075個編碼基因位點(總長度~817,000 bp),數據集2包含了1,948個非編碼基因位點(總長度~111,400 bp)。最終這兩個數據集推斷出了高度相似且解析度良好的游蛇科系統發育樹,其中有85%的節點具有>95%的自展值。上述實驗測試結果表明,FLc-Capture是一種靈活、快速、經濟的目標序列捕獲方法,它可以同時收集編碼和非編碼的系統基因組學數據集,特別有助于研究生命之樹中的困難進化問題。
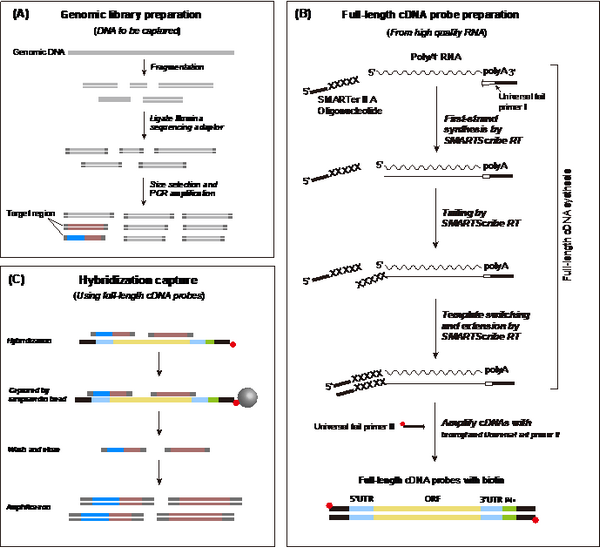
圖1:FLc-Capture的實驗流程

圖2:FLc-Capture的數據分析流程
近日,該成果以“Simultaneously Collecting Coding and Noncoding Phylogenomic Data Using Homemade Full-length cDNA Probes, Tested by Resolving the High-level Relationships of Colubridae”為題發表在學術期刊Frontiers in Ecology and Evolution上。張鵬教授為該項工作的通訊作者,李佳璇博士后為該文第一作者。該研究得到了國家自然科學基金等項目資助。
張鵬教授團隊近年來在基于自制探針獲取分子系統學數據的方法學研究上佳績不斷,除了FLc-Capture方法外,該團隊還開發了以PCR技術為核心的擴增子捕獲技術和適用于解析低階元系統發育關系的AFLP基因組捕獲技術等。這些數據收集策略的上手難度并不高,一般的分子生物學實驗室都可以嘗試,一旦掌握這些技術后,實驗室便能夠自主性、規模性地開展相關的分子系統學研究。