
三代測序技術(PacBio和Oxford Nanopore)可解決基因組重復區域的組裝難題,提高基因組完整性,已成為發育、再生、腫瘤和其它疾病過程中細胞基因組組裝的主流技術。其中,納米孔(Nanopore)測序技術的迅速發展更使得測序成本顯著降低,并且由于其可實現超長讀長(高達1Mbp),在復雜基因組組裝中具有天然優勢。然而,目前Nanopore的測序錯誤分布廣泛(10-30%,圖1A),存在高錯誤局部區域(1000bp中存在50%測序錯誤,圖1B),并且高錯誤局部區域的發生隨著測序讀長增加而顯著增加(圖1C),從而導致超長文庫數據中20-30%的序列存在高錯誤區域。現有的錯誤校正軟件只能通過裁剪的方式剔除高錯誤局部區域,顯著降低了Nanopore序列完整性和組裝完整性。
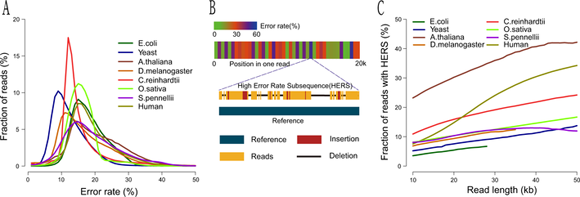
圖 1 Nanopore測序錯誤分布特征
我校中山眼科中心肖傳樂/劉奕志團隊和中南大學王建新團隊于2021年1月4日在Nature Communications雜志上聯合發表題為“Efficient assembly of Nanopore reads via highly accurate and intact error correction”的研究論文,提出了Nanopore漸進式校正組裝模型,開發了相應軟件NECAT,應用于組裝高完整度的視網膜母細胞瘤基因組,并發現了多個結構變異位點。
研究者提出了漸進式序列校正策略,首先選擇高精度的序列校正錯誤率的區域(圖2B),之后優選校正后高精度序列校正高錯誤局部區域,從而保證了序列校正速度和完整性(圖2C);另外,研究者還提出漸進式組裝策略,通過校正后高精度的序列組裝基因組骨架(圖2D),之后通過原始序列提升基因組完整度(圖2E),從而保證基因組組裝結果的正確性和完整性。研究者將上述模型開發了NECAT軟件,開放給國內外其它科研人員,進行長達1年的體驗提升。
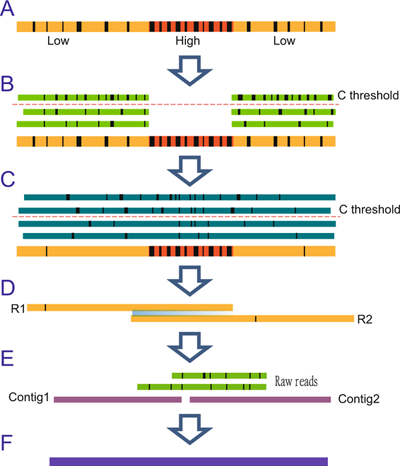
圖2 NECAT校正組裝流程圖
隨后,研究者收集了多種模式生物Nanopore數據集進行性能測試,結果表明:NECAT校正后序列平均精度可達95-98%,可恢復原始數據中99%的高錯誤局部區域(HERS),從而保留了序列長度完整性(表1);NECAT組裝完整性明顯高于同類校正組裝軟件,且組裝錯誤量顯著低于同類軟件。另外,研究者將NECAT校正結果與多個組裝軟件結合使用發現:NECAT校正結果顯著提高其它Nanopore組裝軟件的組裝質量。
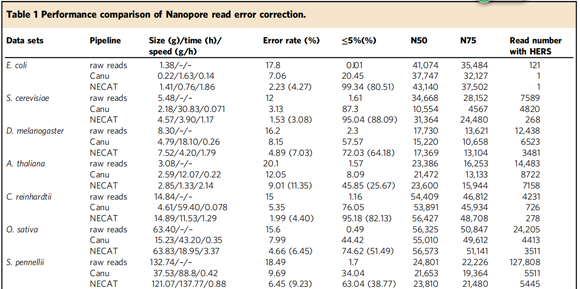
表1 NECAT序列錯誤校正性能評估
最后,研究者完成了視網膜母細胞瘤Nanopore測序,并應用NECAT組裝出了完整度較高母細胞瘤癌癥基因組,通過組裝結果發現了很多高精度結構變異(SV)位點,其很多位點都與目前實驗報道和功能預測相符(圖3)。與原始數據SV檢測方法相比,NECAT組裝結果檢測SV精度顯著高于目前SV檢測方法。上述結果表明,通過NECAT序列校正,顯著降低高錯誤區域所造成的SV假陽性結果。
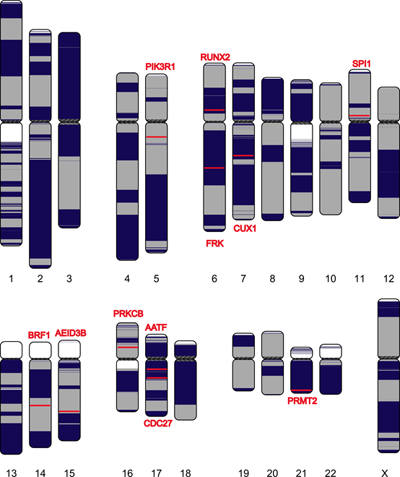
圖3 視網膜母細胞瘤基因組染色體圖譜及SV位點
綜上所述,本研究提出的漸進式校正組裝方法可以有效解決了Nanopore復雜測序錯誤問題,顯著提高了Nanopore數據組裝完整性、正確性和數據利用率。另外,通過NECAT序列校正,可以有效降低高錯誤區域SV的假陽性。