
中大新聞網訊(通訊員胡友金、唐艷麗)視網膜神經發育和再生的分子機制復雜,涉及DNA變異、表觀遺傳修飾、RNA轉錄和剪接等,多組學整合分析被認為是揭示神經干細胞譜系分化和再生分子機制的有效手段。由于神經發育和再生過程中多個神經譜系交錯在一起,如何準確地分辨不同譜系并系統描繪其多維分子特征的動態變化,是精準解析其分子機制的關鍵所在。近年來,單細胞多組學測序技術的發展,為分析神經元發育和再生復雜分子機制提供了有力方法。然而,RNA可變剪接作為調控基因功能的重要環節,尚缺乏有效的單細胞多組學關聯分析方法。
2024年9月13日,中山大學中山眼科中心胡友金教授團隊在Nature Communications雜志在線發表了題為“Simultaneous profiling of RNA isoforms and chromatin accessibility of single cells of human retinal organoids”的研究論文,報道了一種名為scRICA-seq (single-cell RNA isoform and chromatin accessibility sequencing) 的單細胞多組學測序方法,該技術能夠同時檢測單細胞水平染色質可及性、RNA表達,以及 isoform全長結構,實現了單個細胞水平的表觀遺傳學、轉錄與RNA剪接的整合分析,為揭示視網膜神經元發育和再生的新機制提供技術支撐。
胡友金團隊長期專注于單細胞測序技術的創新開發。早在2020年,該團隊已在《Nature Communications》上發表scRCAT-seq技術,用于RNA isoform的檢測。此次,團隊進一步改進原有方法,發展了scRCAT-seq2全長轉錄組測序技術,并結合10x Genomics的Chromium Next GEM Single Cell Multiome ATAC + Gene Expression平臺,實現了染色質可及性與RNA isoform同時檢測的多組學方法。其中,scRCAT-seq2的核心技術是通過單分子成環技術處理cDNA,并將覆蓋TSS,TES和exon的測序序列進行同一標簽標記,并組裝構建全長cDNA序列。(圖1)
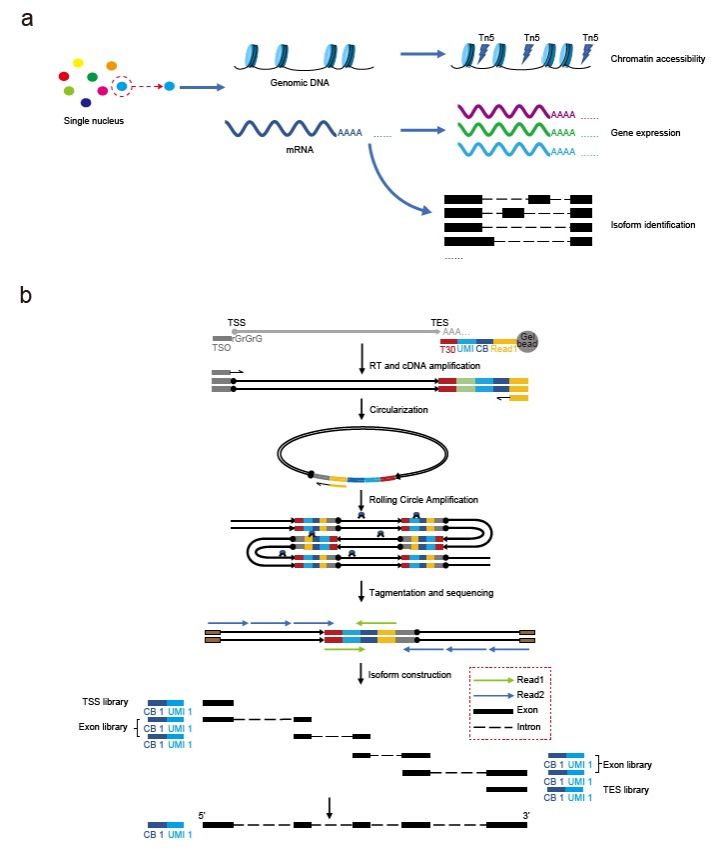
圖1:scRICA-seq和scRCAT-seq2的示意圖
研究者將scRCAT-seq2在低通量與高通量條件下與多種三代測序技術(如SCAN-seq2、scISOr-seq、HIT-scISO-seq等)進行對比分析,結果顯示scRCAT-seq2在成本效益和檢測靈敏度方面均具有顯著優勢。(圖2)
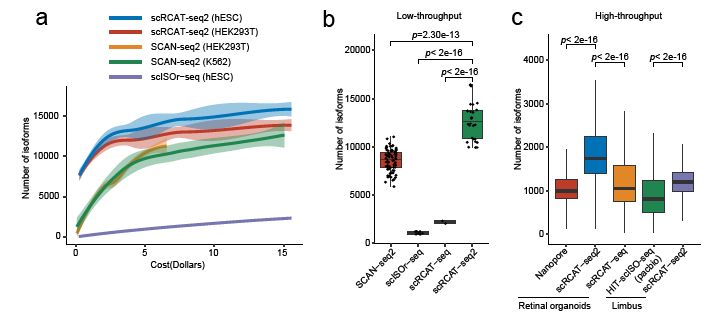
圖2:scRCAT-seq2與其他方法比較分析圖
研究者將scRICA-seq應用于人類視網膜發育過程,構建了人類視網膜類器官的單細胞多組學圖譜。基于單細胞異構體的聚類分析,確定了六個細胞簇,對應于六種典型的視網膜早期神經元,包括視網膜祖細胞(RPCs)、視網膜神經節細胞(RGCs)、無長突細胞/水平細胞(AC/HCs)、視錐細胞前體(PR precursors)、神經生成RPCs(neurogenic RPCs)以及視錐細胞(cones),其結果與scRNA-seq技術所得數據高度一致。(圖3)

圖3:基于scRICA-seq的人類視網膜類器官的多組學單細胞圖譜
進一步,作者研究了RPC分化為cone過程中RNA isoform的動態選擇模式,發現cone和RPC之間共有2292種差異表達的isoform,其中1143種isoform在cone中高度表達,1149種在RPC中高度表達。通過轉錄因子motif分析,研究者預測了589個可能與這些動態調節的剪接位點結合的轉錄因子。在預測的轉錄因子中包含了11種先前報道的參與調節RNA剪接的轉錄因子,包括NEUROD1、SP1和KLF4等,同時還包含了已知的細胞命運決定因子,如NEUROD1、ASCL1和THRB等。這提示,剪接位點染色質可及性的動態變化與RNA剪接可能協同調控細胞命運決定(圖4)。
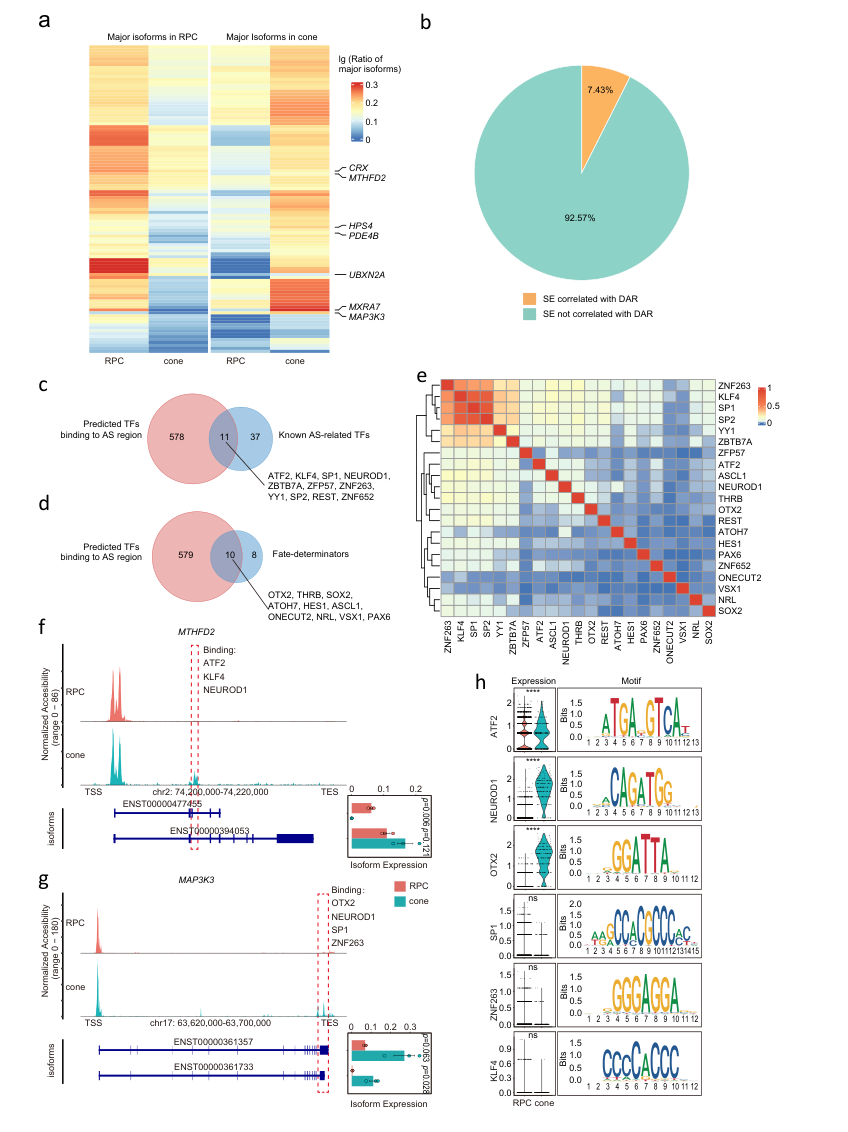
圖4:RPC發育過程中RNA isoform選擇與染色質可及性和TFs的相關性
綜上所述,scRICA-seq在單細胞水平實現了表觀遺傳學、RNA轉錄和剪接的整合分析,發現染色質可及性和命運決定因子協同調控下游基因的表達和轉錄剪接,為闡明視網膜神經元發育和命運決定的機制提供了新思路。
本研究由中山大學中山眼科中心胡友金教授課題組完成。中山眼科中心張淑堯(研究助理)、肖玉華(博士研究生)、莫新枝(研究助理)為共同第一作者,胡友金教授為唯一通訊作者。中山大學中山眼科中心、眼病防治全國重點實驗室為第一作者單位和通訊作者單位。
論文鏈接:
https://www.nature.com/articles/s41467-024-52335-0