
中大新聞網訊(通訊員熊麗娜)在成熟的多細胞生物體內,具有特定功能的細胞群通常源自于一組祖先細胞。細胞群體的譜系多樣性是由這些祖細胞的數量決定的,因此估計祖先細胞群體數量是發育生物學的重要研究問題之一。而傳統的研究手段主要依賴于組織特異性的啟動子介導報告基因的表達,進而對目標細胞群體進行計數。這一類研究手段的可靠性高度依賴于啟動子的選擇,其中,啟動子的組織特異性與時空表達動態都會使估計結果產生偏差。
2024年2月20日,中山大學生命科學學院賀雄雷課題組在Nature Methods上發表了一篇題為A statistical method for quantifying progenitor cells reveals incipient cell fate commitments的研究文章。該研究基于近年來逐漸成熟的細胞譜系追蹤技術構建的發育細胞譜系樹 (developmental cell phylogeny),結合群體遺傳學的經典溯祖理論 (coalescent theory) 思想,建立了一種估計祖先細胞群體大小的統計方法TarCA (targeting coalescent analysis),以此來研究胚胎發育過程中細胞群體的動態變化。
給定一顆包含多種細胞類型的細胞譜系樹,它的末端節點代表了被采樣的細胞,而中間節點則表示祖先細胞。如果一個中間節點的所有子代細胞均為相同的細胞類型,研究人員將其定義為細胞類型特異的單系分支 (monophyletic clade)。隨后,研究人員根據單系分支的大小分布計算出隨機兩個細胞均來自同一單系分支的概率,而該類型細胞的祖先細胞群體數量即為該概率的倒數(圖1)。
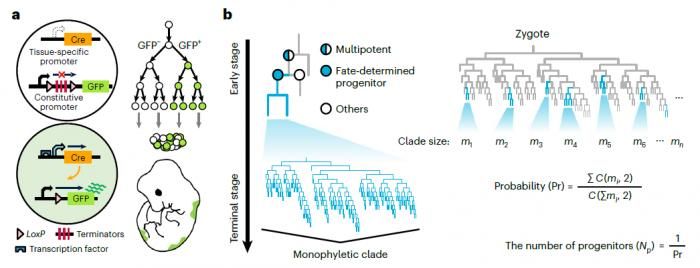
圖1 估計祖先細胞數目的傳統方法與TarCA
通過計算機模擬數據,該研究發現,與對單系分支進行直接計數的方法相比,TarCA對采樣率要求更低,并且在實驗噪音存在時仍能夠快速收斂。這是因為在TarCA算法中,子代細胞數量較少的祖先細胞權重較低,從而TarCA能穩定地估計出對子代細胞群體做出主要貢獻的祖先細胞數量。隨后,研究人員在線蟲、果蠅與小鼠的細胞譜系數據中,驗證了TarCA在處理真實生物學數據中的可靠性。以小鼠為例,研究人員發現不同組織的祖先細胞群體數量在個體間高度一致。另外,除采樣獲得的終末細胞類型外,TarCA還能對發育初期的胚胎結構(如三胚層及胚外組織等)的祖先細胞群體數量進行估計,進一步證明了TarCA的可靠性與泛用性。
最后,研究人員還將TarCA拓展至早期細胞命運決定過程的研究中。在細胞命運決定的初期,群體內部僅有小部分驅動基因的表達發生了變化,在全轉錄組水平依然保持穩定;隨著細胞命運決定的進行,細胞群體內逐漸產生全轉錄組水平的表達差異。如何在基因水平,而非全轉錄組水平,對基因表達在細胞群體中的異質性進行檢測,是研究早期細胞命運決定過程的關鍵難題(圖2)。為此,該研究使用TarCA算法對基因表達水平在細胞譜系樹上的異質性進行了評估,識別出了表達水平與細胞分裂歷史事件偶聯的基因(lineage-specific upregulated genes, LUGs)。以小鼠早期胚胎的腸管細胞為例,研究人員篩選出了155個LUGs,其中包含眾多標記前、中、后腸的明星基因。根據LUGs在不同細胞中的表達情況,研究人員進一步構建出小鼠腸管向下游器官分化過程的層級模型。該模型指出,胰腺、小腸與結腸間存在共享的祖先狀態,與體外定向分化實驗結果一致。
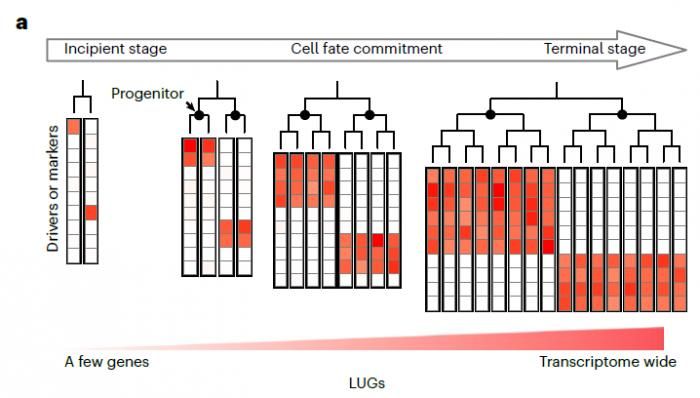
圖2 定義細胞命運分配過程中的LUGs
總的來說,該研究開發的TarCA算法可對任意細胞群體估計祖先細胞數目,而無需事先獲得祖細胞的先驗信息,也能推算少數細胞中的驅動基因,以此來預測早期細胞命運的分化傾向。這些發現說明TarCA能促進包括人類在內的所有復雜生物組織的譜系多樣性的量化,對其建立定量模型至關重要,可以幫助人們解決個體發育可塑性和衰老相關的組織功能退化的問題。這項工作是經典進化生物學與現代細胞發育生物學融合的一個典范。
中山大學生命科學學院賀雄雷教授為該論文的通訊作者,課題組博士后鄧善俊與博士畢業生龔晗為共同第一作者。該研究得到了國家重點研發計劃與國家自然科學基金的資助。
論文鏈接:https://www.nature.com/articles/s41592-024-02189-7
TarCA算法工具鏈接:https://github.com/shadowdeng1994/TarCA